Resistens kan i noen tilfeller også bestemmes direkte på gennivå ved blant annet PCR-baserte metoder og DNA-sekvensering, for å påvise resistensgener. Fordi resistens ofte kodes genetisk, kan avlesning av baserekkefølgen i DNA ved sekvensering gi oss svar på om kjente resistensfaktorer er til stede.
Ved PCR brukes primere som er komplementære og binder seg (hybridiserer) til starten og slutten av genet man ønsker å kopiere og påvise. Et eksempel er fremgangsmåten for å undersøke forekomst av methicillinresistente stammer av bakterien Staphylococcus aureus (MRSA). Det fins ett spesifikt gen, mecA-genet, som gjør denne bakterien resistent mot methicillin. Det er mulig å bruke PCR for å påvise mecA-genet.
De molekylære teknikkene er ikke avhengige av dyrkning, og er derfor spesielt nyttige for resistensbestemmelse av mikroorganismer som vanskelig lar seg dyrke i laboratoriet eller som vokser sakte, som tuberkelbakterien (Mycobacterium tuberculosis).
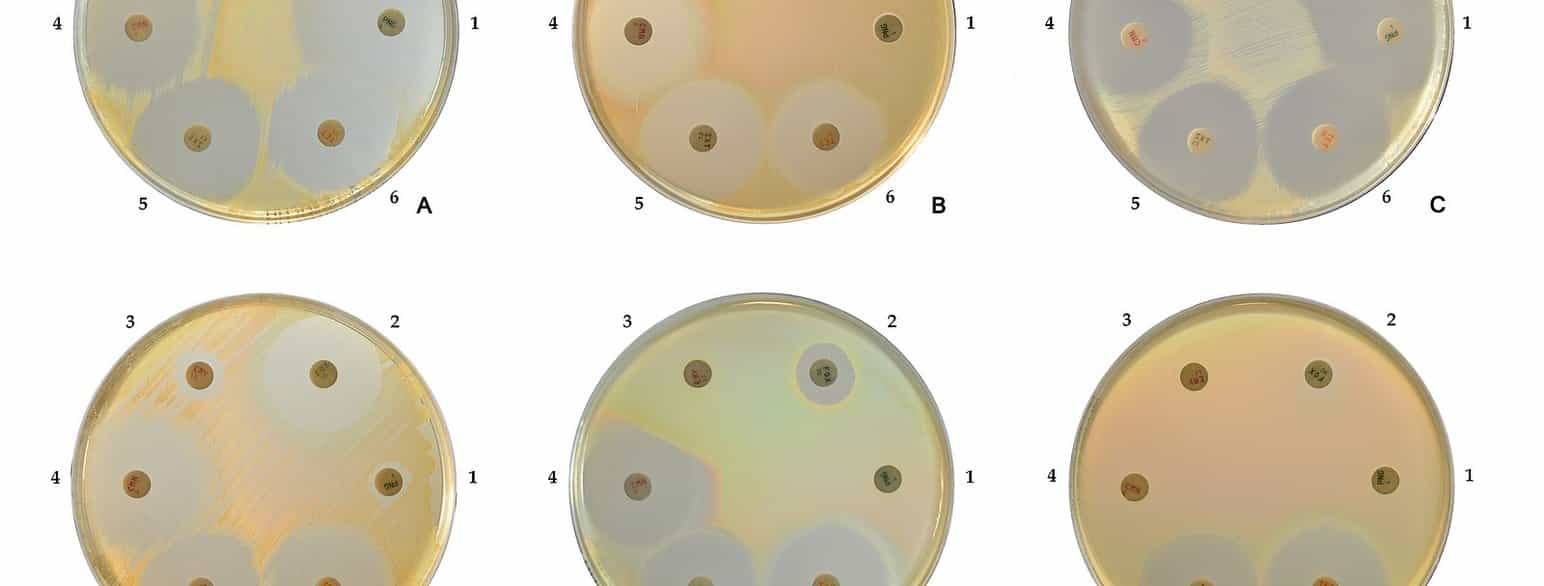
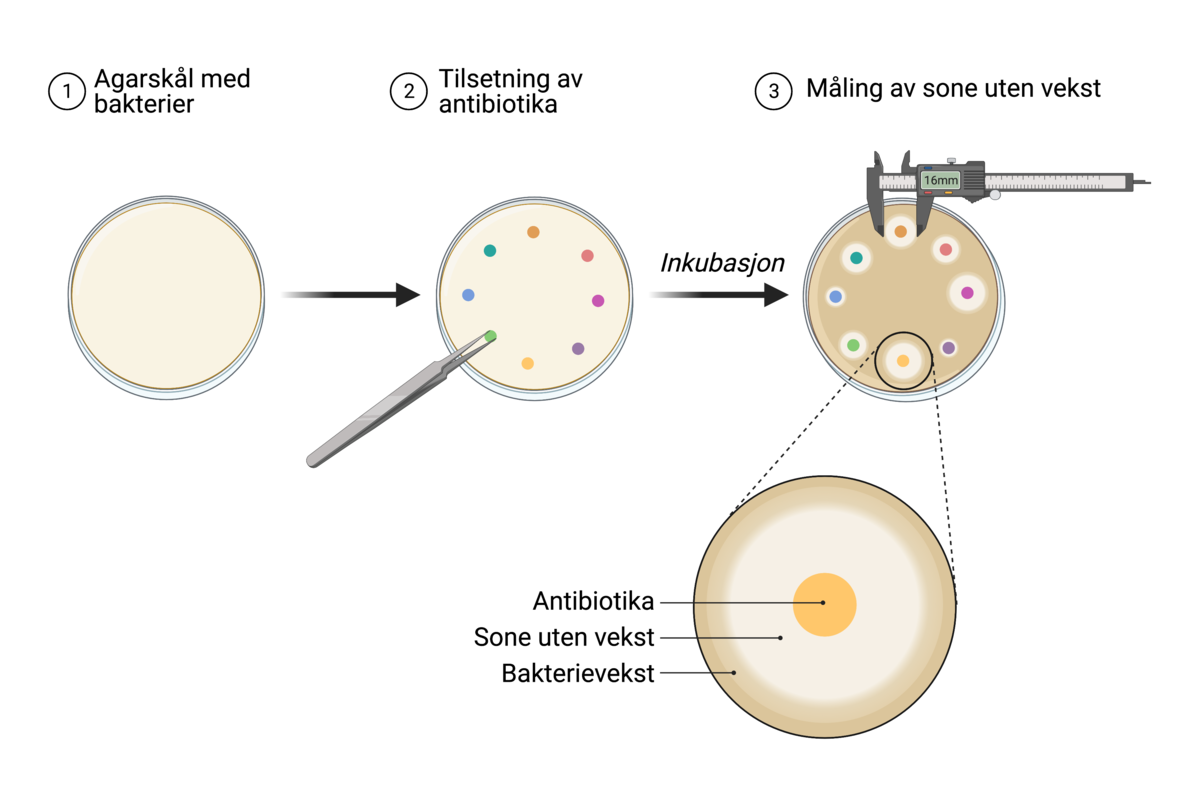
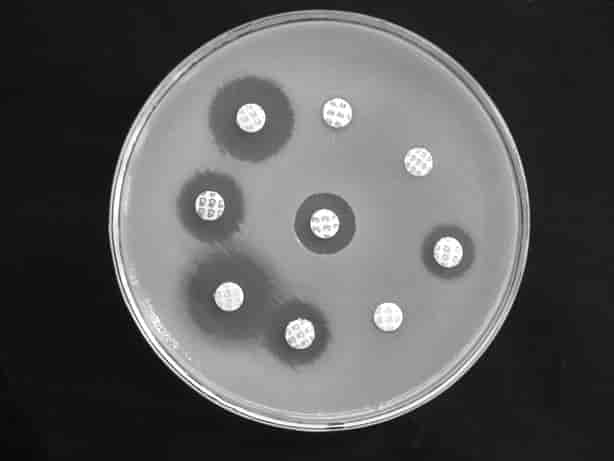
Antibiotikaresistens kan imidlertid avhenge av flere faktorer enn tilstedeværelse av gener som koder for resistens. Selv om molekylære metoder og bruk av sekvensdata er stadig under utvikling, er man derfor i mange tilfeller fortsatt avhengig av å dyrke mikroorganismene ved resistensbestemmelse.



Kommentarer
Kommentarer til artikkelen blir synlig for alle. Ikke skriv inn sensitive opplysninger, for eksempel helseopplysninger. Fagansvarlig eller redaktør svarer når de kan. Det kan ta tid før du får svar.
Du må være logget inn for å kommentere.